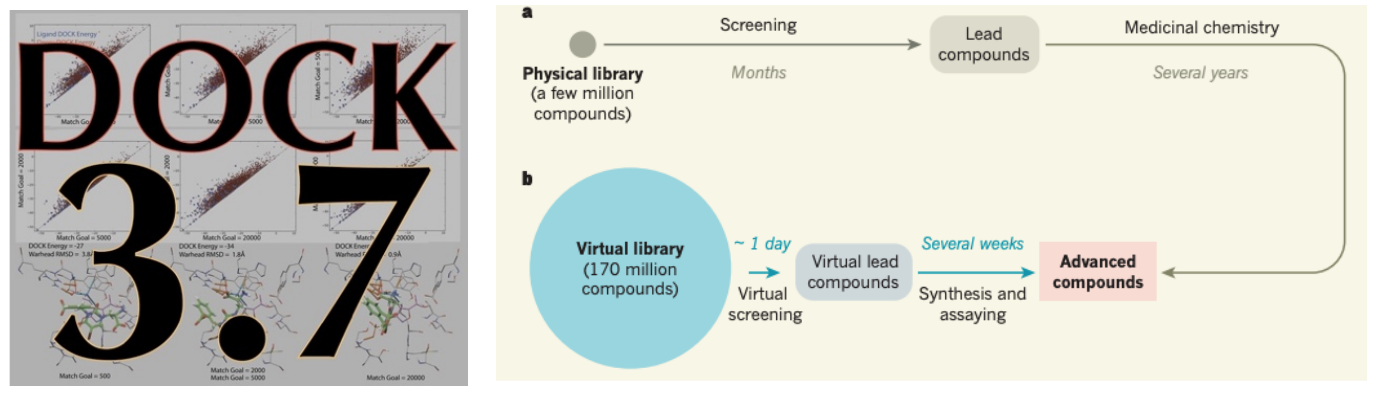
分子对接(DOCK)是基于蛋白三维结构进行化合物虚拟筛选的广泛使用的计算工具。目前针对超大规模化合物库进行分子对接,发现全新骨架的新分子是计算化学研究的热点[1]。随着可合成虚拟库中分子数量的激增,对常规的基于对接软件来说,计算时间和成本就成了瓶颈。例如,以10秒/分子的速度对接100亿个化合物,在单个CPU核上需要跑3000年以上,如果上云计算,需要的花费可能超过80万美元。所以在保持对接精度前提下,对现有对接软件进行算法的高性能优化,大幅度减少计算时间的研究工作就显得非常具有实际应用价值。
“神威·太湖之光”超级计算机是国家863计划信息技术领域重大项目支持的课题,它也是全球第一台性能超过十亿亿次的计算器,并且采用国产高性能众核处理器SW26010构建[2]。直到今天,它依然在2022年上半年全球超级计算机Top500排名世界第六位。为了充分利用无锡超算的强大计算性能,山东大学软件学院刘卫国教授团队,北京清华大学计算机科学与技术系杨广文教授团队,北京生命科学研究所黄牛实验室团队和康迈迪森(北京)医药科技有限公司团队一起对分子对接软件UCSF DOCK3.7在“神威·太湖之光”超级计算机上进行重新设计和优化。
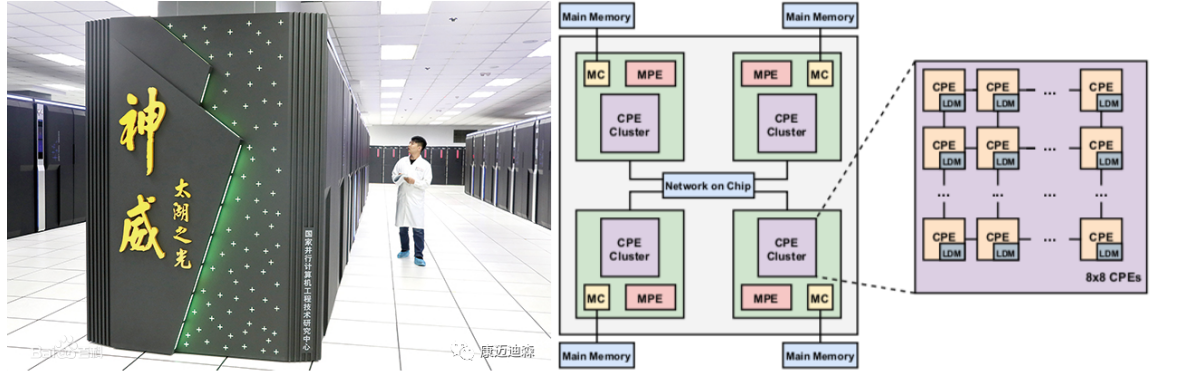
我们在“神威·太湖之光”超级计算机上的性能加速策略主要包括:1)为了避免负载不平衡的影响,采用了生产者-消费者策略,将输入/输出进行高性能优化;2)新的二进制文件格式,以取代原来的用于小分子三维结构存储的文件格式;3)对现有分子对接程序中最耗时的小分子与蛋白方向匹配的算法进行了优化;4)重新对代码优化,并压缩了内存的使用,将数据存储在快速的本地设备存储器(LDM)中;5)提出了一些特定架构的优化方案。实现了异步数据传输和计算的矢量化,以充分利用SW26010处理器的优势。
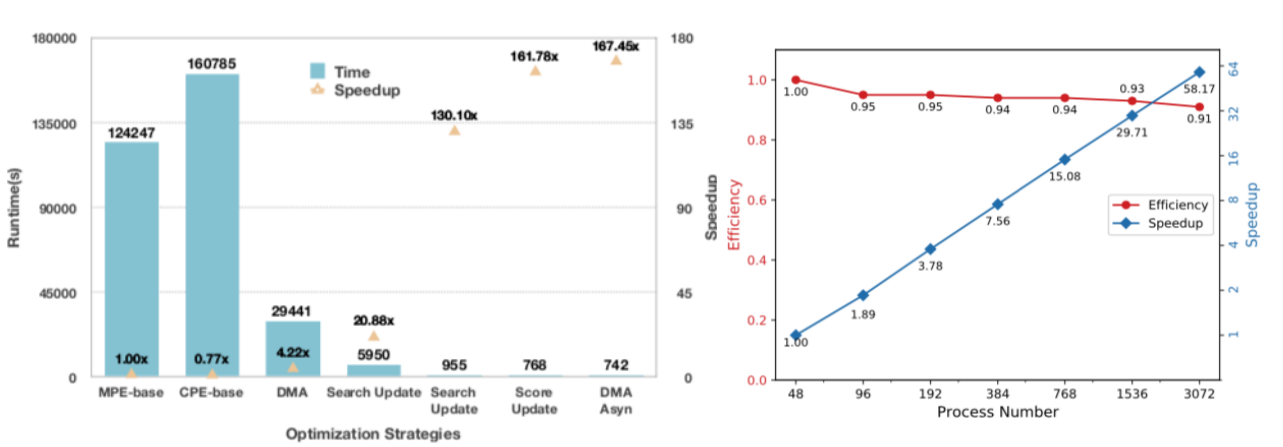
最终,我们的整体优化策略实现了高达167倍的速度提升,同时展现了在下一代Sunway超级计算机上对数十万个异构核心的强大可扩展性[3]。未来康迈迪森将进一步开发模块化的基于合成子(分子量250~350Da的刚性小分子片段)和组合化合物库的新对接算法[4],结合神威系列国产超级计算机的强大的算力,进一步拓展基于蛋白三维结构的虚拟筛选在更大化合物空间的应用,筛选出更多具有开发潜力的苗头化合物,加速早期药物开发,降低成本。
参考文献:
[1]. Lyu, Jiankun, et al. “Ultra-large library docking for discovering new chemotypes.” Nature 566.7743 (2019): 224-229.
[2]. http://www.nsccwx.cn/
[3]. IEEE Transactions on Parallel and Distributed Systems, 2022. 10.1109/TPDS.2022.3194916
[4]. Sadybekov, Arman A., et al. “Synthon-based ligand discovery in virtual libraries of over 11 billion compounds.” Nature 601.7893 (2022): 452-459.